WGCNA
AI-摘要
海底捞里没有鱼 GPT
AI初始化中...
介绍自己 🙈
生成本文简介 👋
推荐相关文章 📖
前往主页 🏠
前往爱发电购买
WGCNA
泡泡WGCNA 入门
- 权重(weghted):基因之间不仅仅是相关与否,还记录着它们的相关性数值,数值就是基因之间的联系的权重(相关性)。
- 模块(module):表达模式相似的基因分为一类,这样的一类基因成为模块。
- Eigengene:基因和样本构成的矩阵。
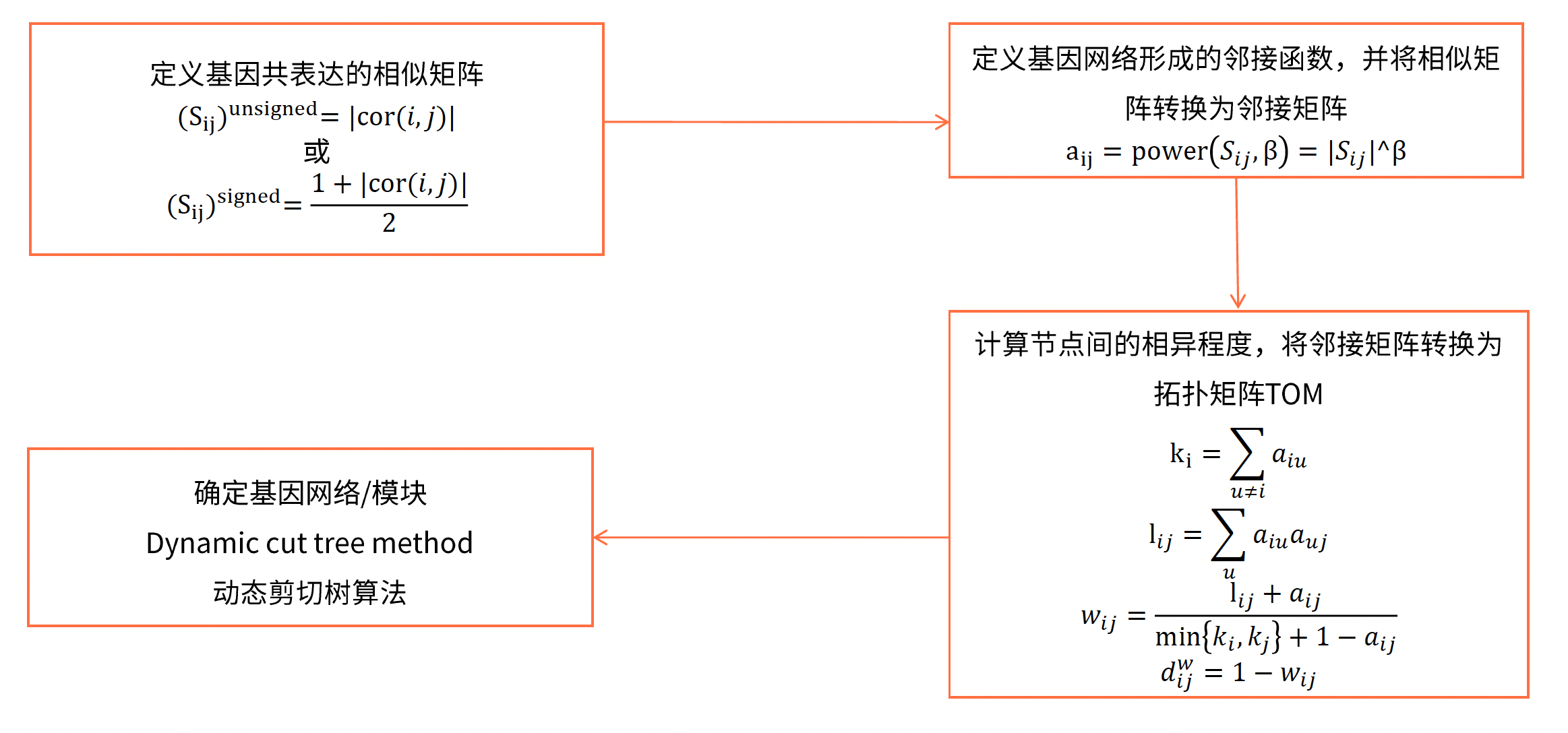
- 邻接矩阵(Adjacency Matrix):是图的一种存储形式,用一个一维数组存放图中所有顶点数据;用一个二维数组存放顶点间关系(边或弧)的数据,这个二维数组称为邻接矩阵;在WGCNA分析里面指的是基因与基因之间的相关性系数矩阵。 如果用了阈值来判断基因相关与否,那么这个邻近矩阵就是0/1矩阵,只记录基因相关与否。但是WGCNA没有用阈值来卡基因的相关性,而是记录了所有基因之间的相关性。
- 拓扑重叠矩阵(TOM,Topological Overlap Matrix):WGNA认为基因之间的简单的相关性不足以计算共表达,所以它利用邻近矩阵,又计算了一个新的邻近矩阵。一般来说,TOM就是WGCNA分析的最终结果,后续的只是对TOM的下游注释。
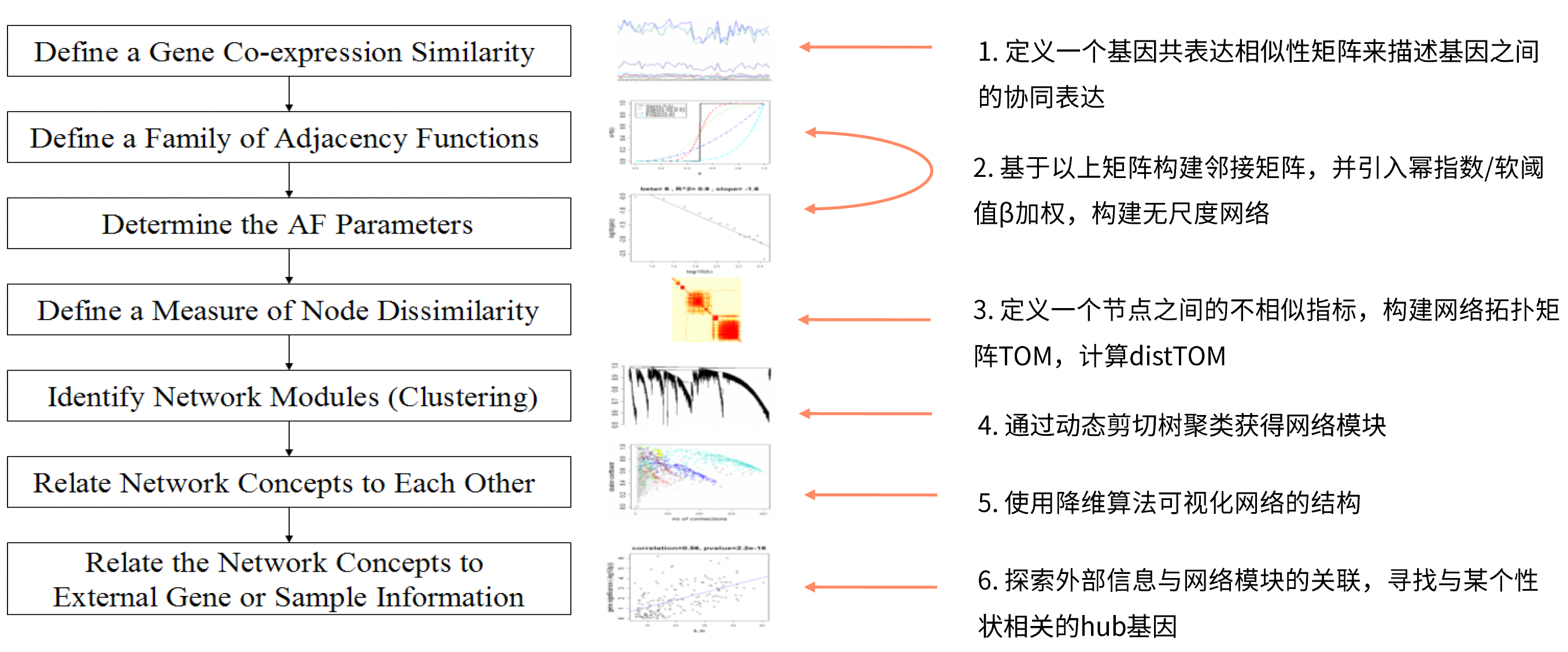
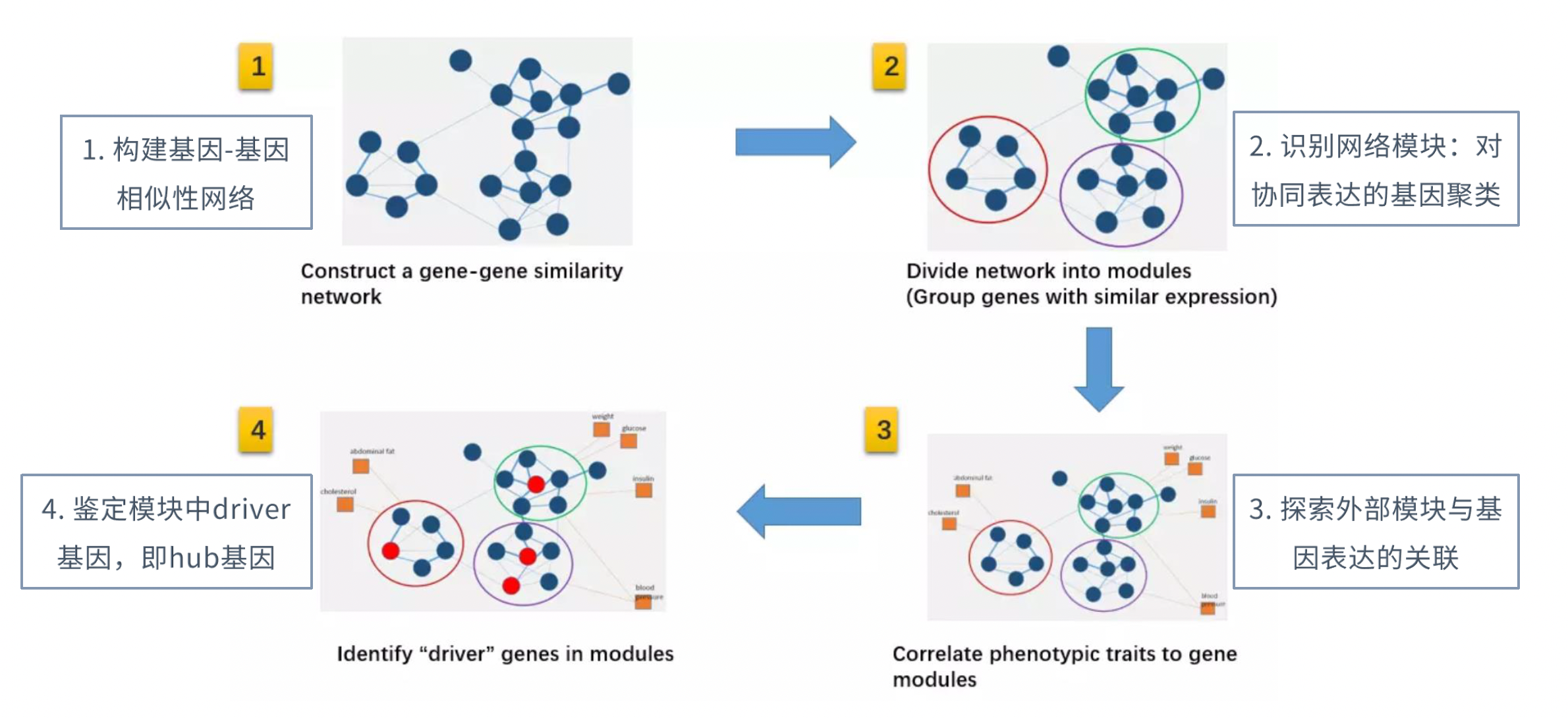
- WGCNA(Weighted Gene Co-expression Network Analysis),即加权基因共表达网络分析。 是一种构建基因共表达网络的常用系统生物学算法,与传统的基因表达网络算法的主要差异在WGCNA采用了软阈值β对表达矩阵进行了加权。
- WGCNA的分析目的在于寻找具有协同表达或共表达的基因组成的网络模块,探索这些基因网络模块与研究的表型或性状之间的关系,寻找与外部信息相关的Hub基因。
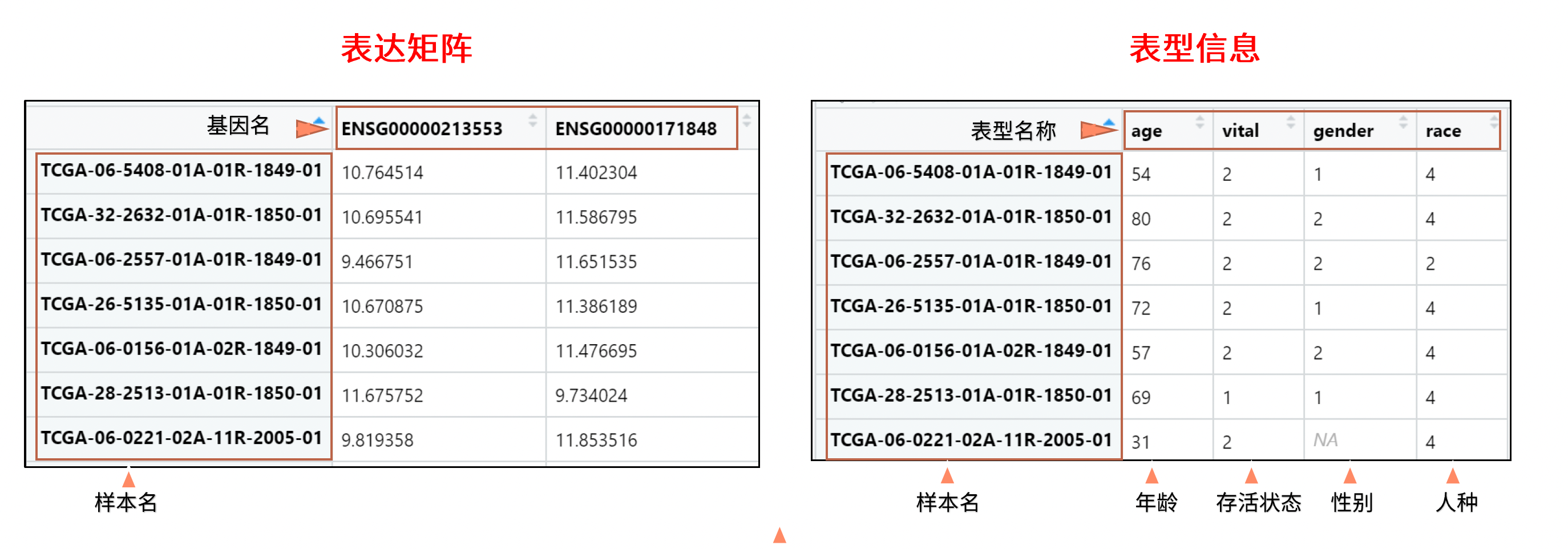
数据要求
- 表达矩阵
- 表型信息
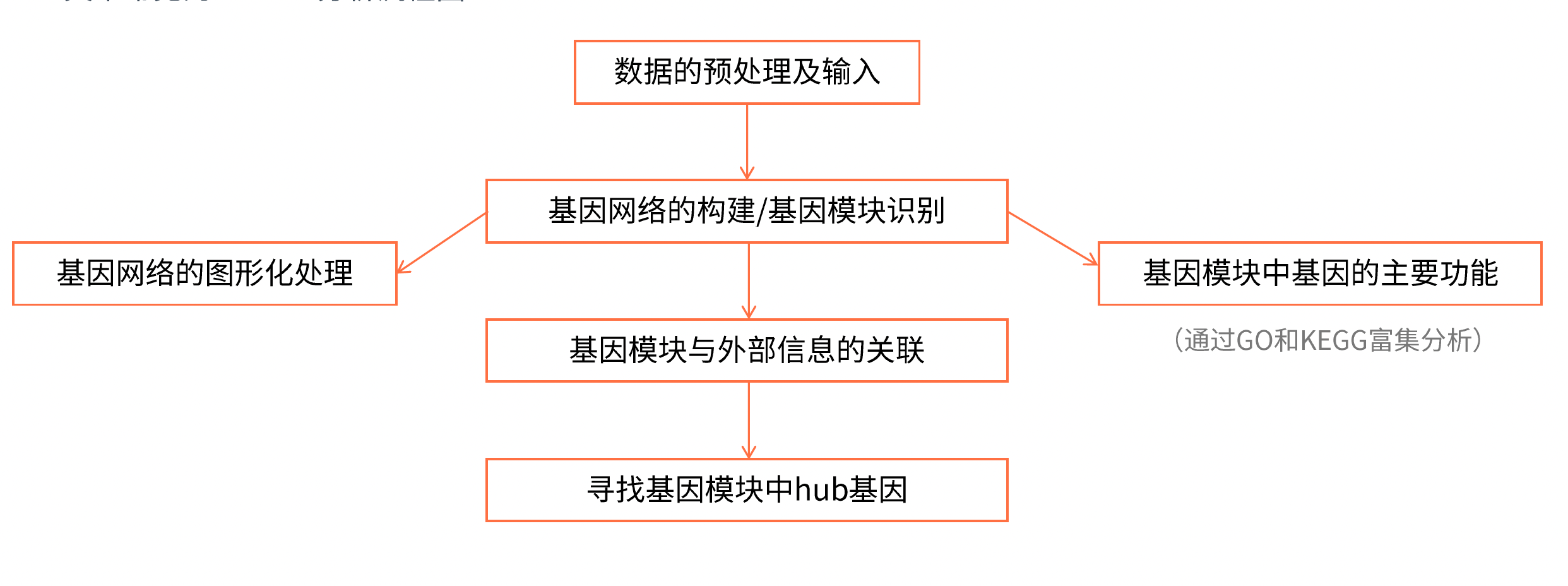
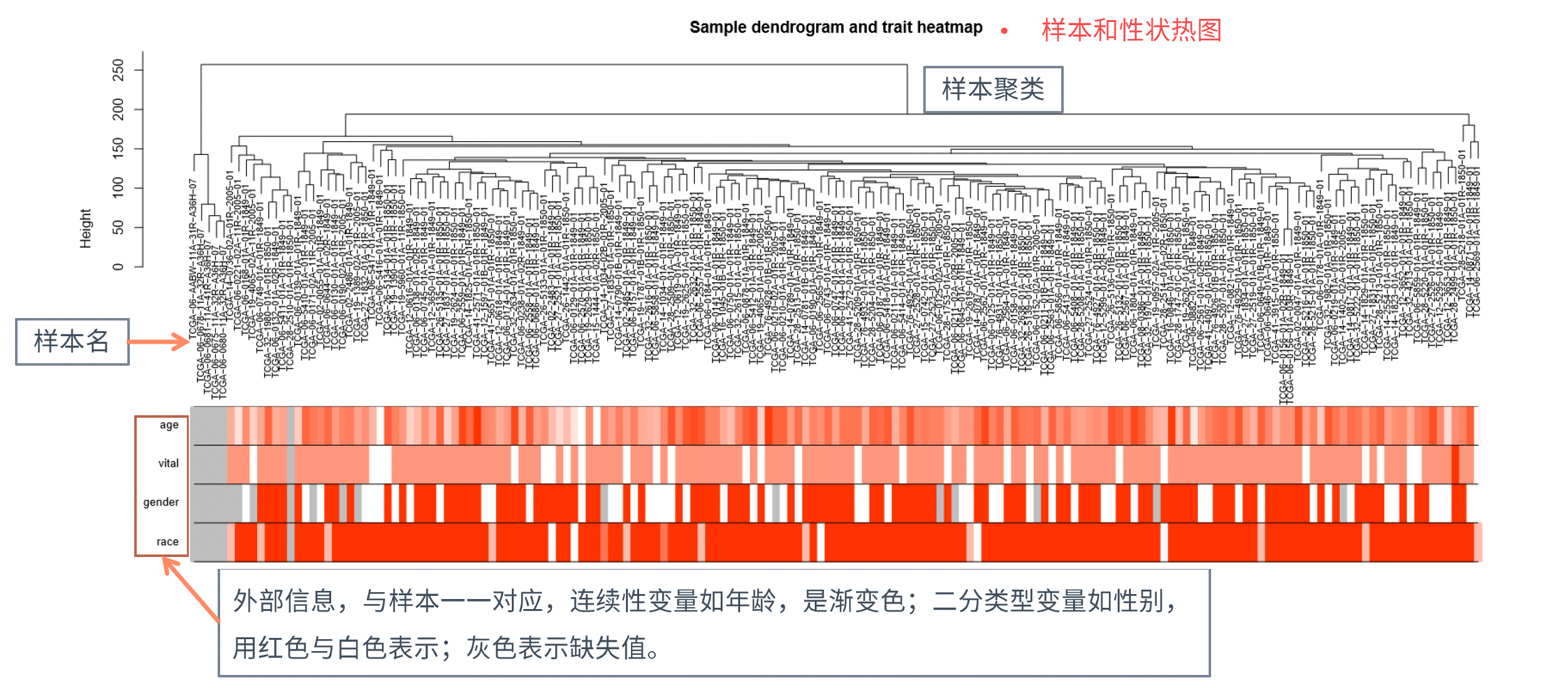
分析手段
R包
- WGCNA包
结果解读
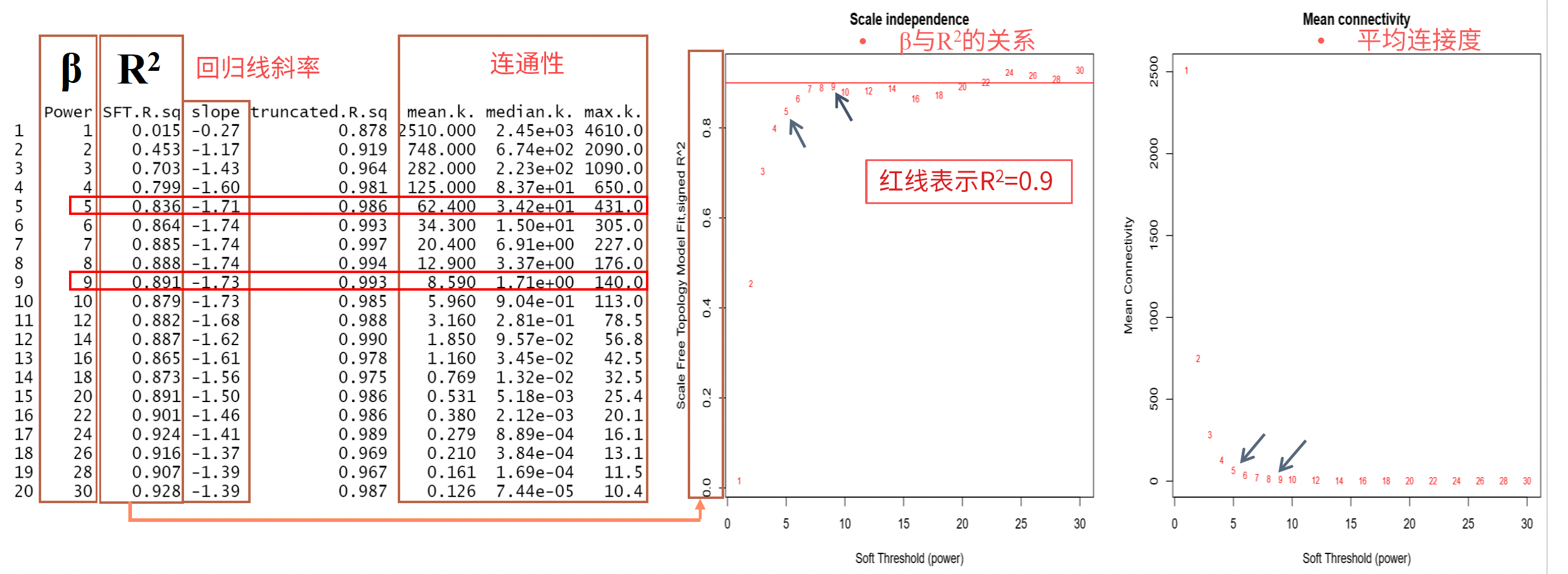
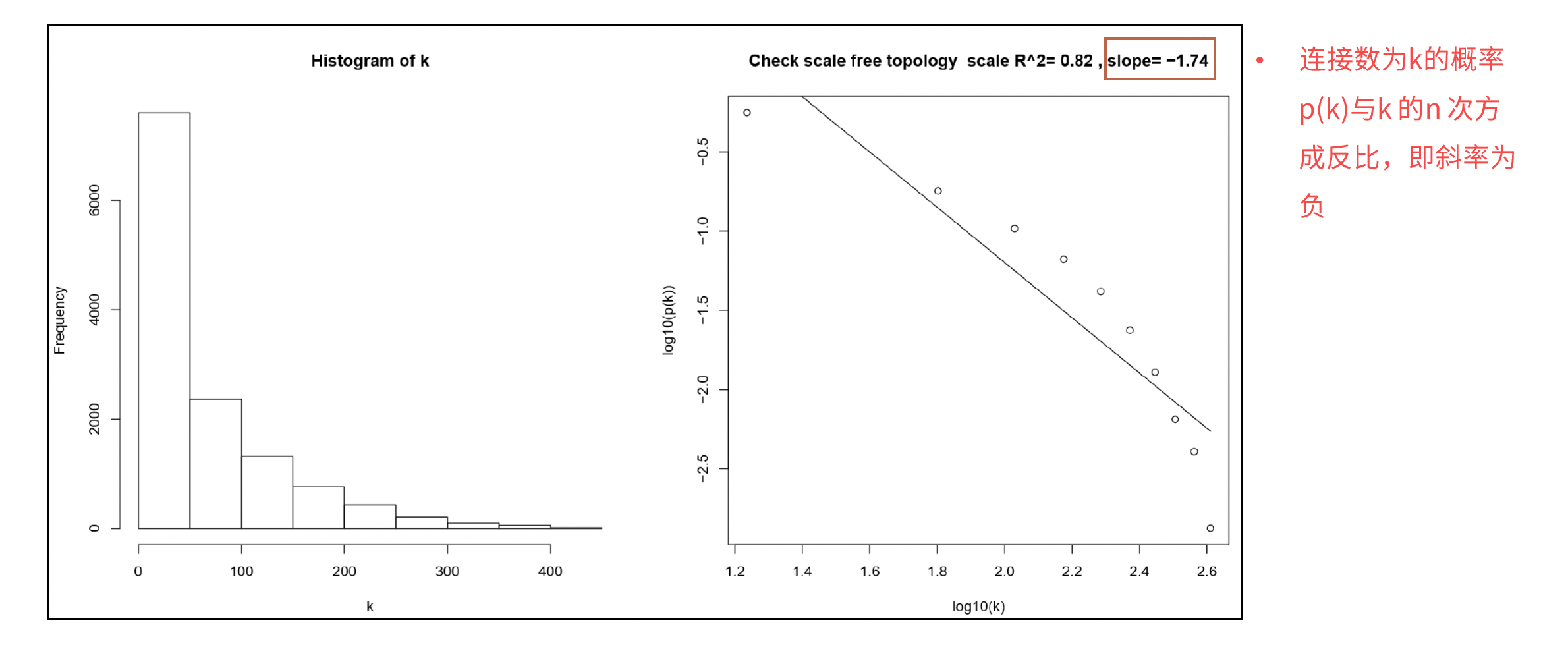
为了衡量两个基因是否具有相似表达模式,一般需要设置阈值来筛选,高于阈值的则认为是相似的。但是假设直接将阈值设为0.8,那么很难说明0.8和0.79两个是有显著差别的。因此,WGCNA分析时采用相关系数加权值,即对基因相关系数取N次幂,使得网络中的基因之间的连接服从无尺度网络分布(scale-freenetworks),这种算法更具生物学意义。
- 如例图所示,随着ß增大,R^2^增大,网络的连通性降低;ß=9时呢,网络的连通性很低,这时我们考虑降低R^2^的选择,选择ß=5。(R^2^最好不要低于0.8)
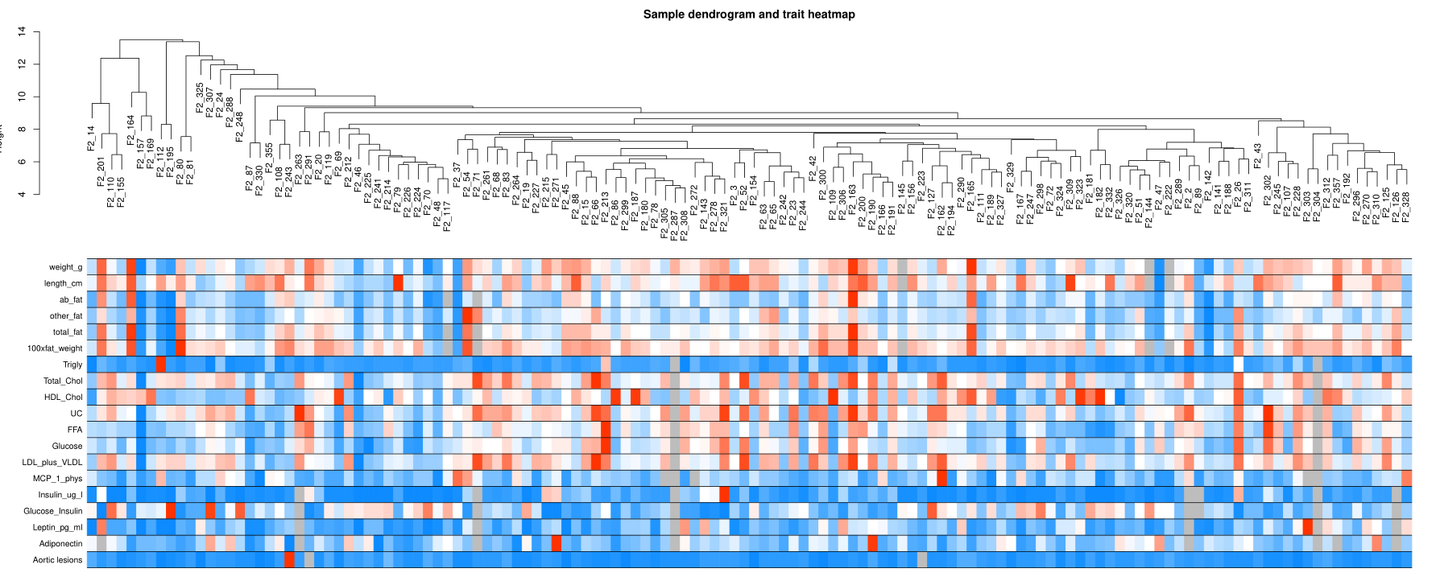
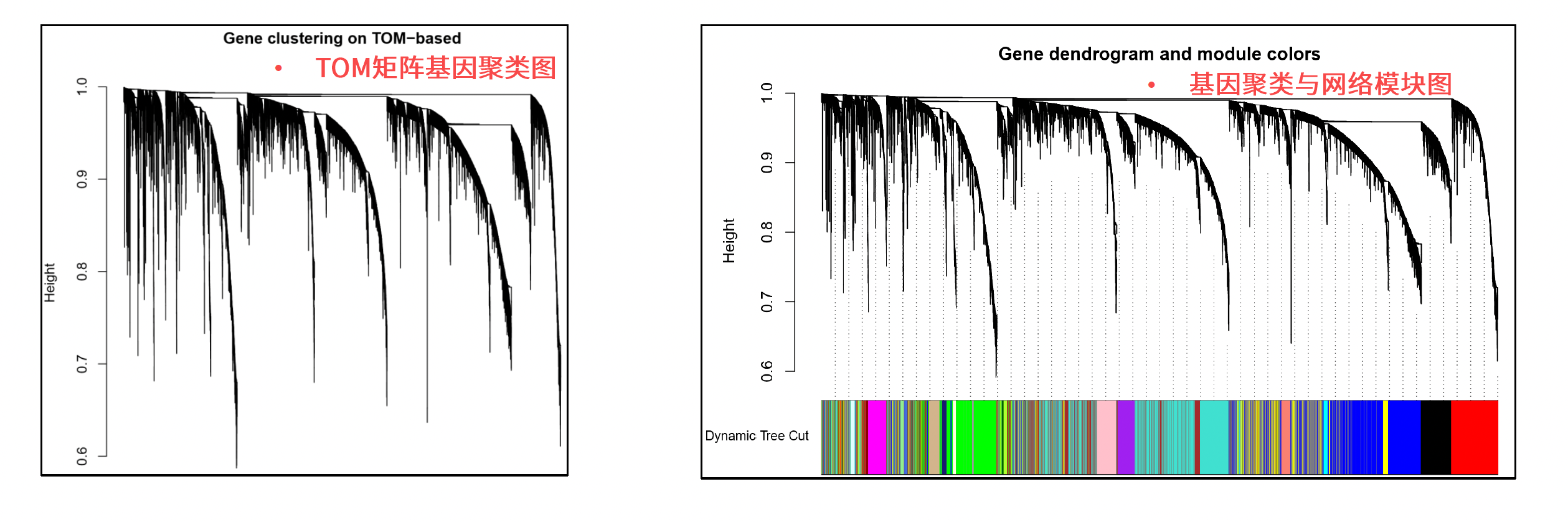
通过基因之间的相关系数构建分层聚类树,聚类树的不同分支代表不同的基因模块,不同颜色代表不同的模块。基于基因的加权相关系数,将基因按照表达模式进行分类,将模式相似的基因归为一个模块。这样就可以将几万个基因通过基因表达模式被分成了几十个模块,是一个提取归纳信息的过程。
- 动态剪切树算法对基因拓扑矩阵进行聚类,初步识别网络模块
- Height表示基因之间的相异性程度,越大表示基因协同表达差异越大,不属于一个网络模块
- 不同颜色用于表示不同的网络模块
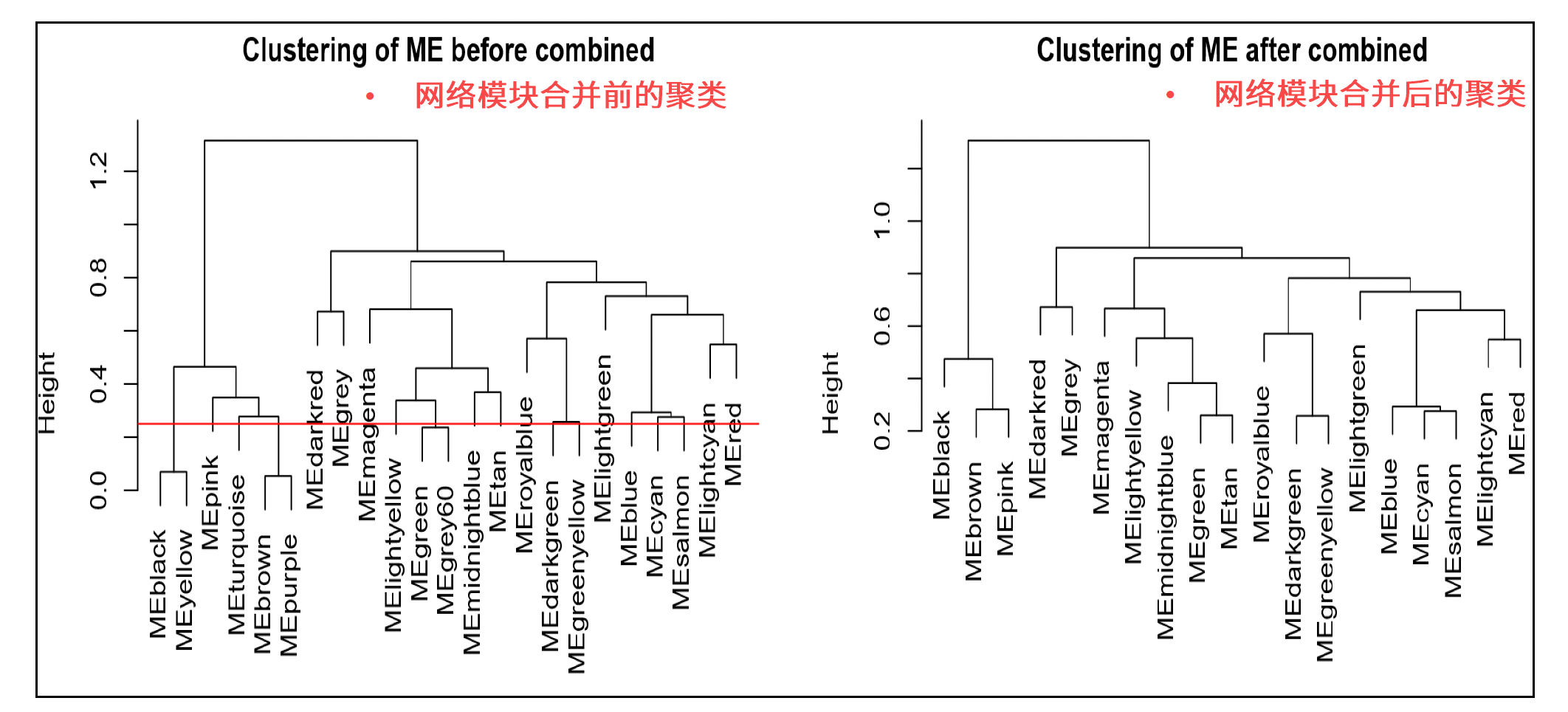
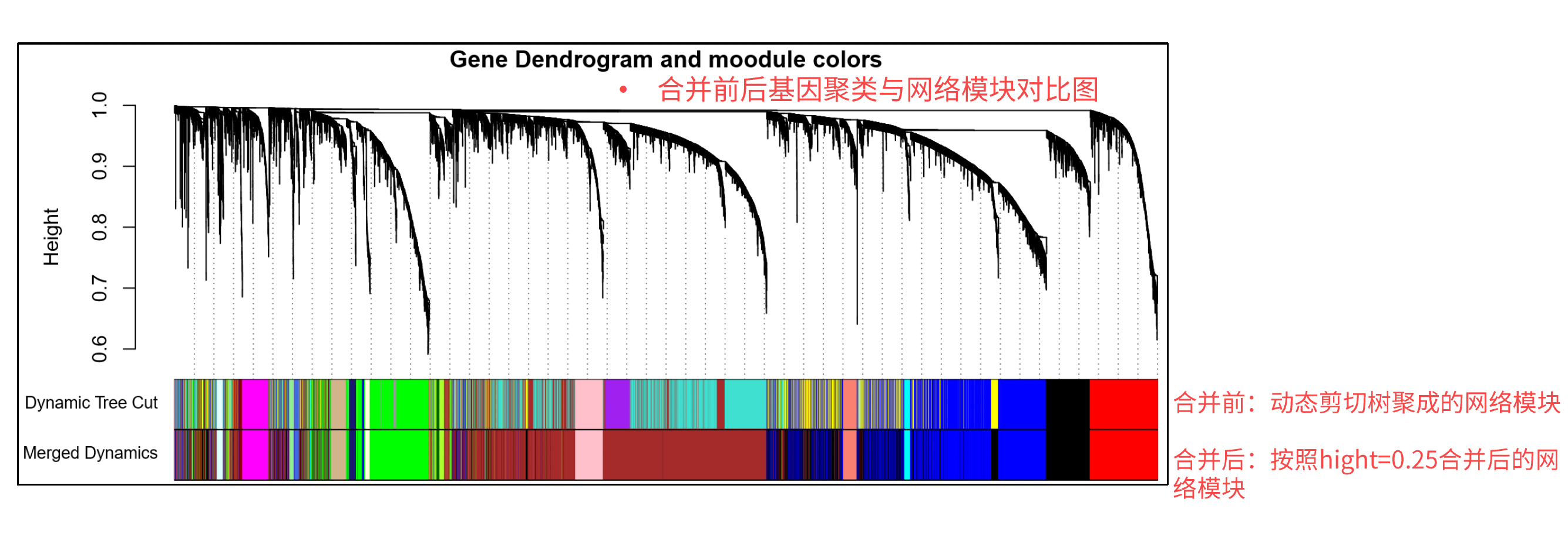
对初步构建的网络模块进行合并, 左图中红线表示0.25,在红线下面表示:这些模块之间的相异程度小于0.25,即相似程度高于0.75,将这些模块合并为一个模块,如右图所示。
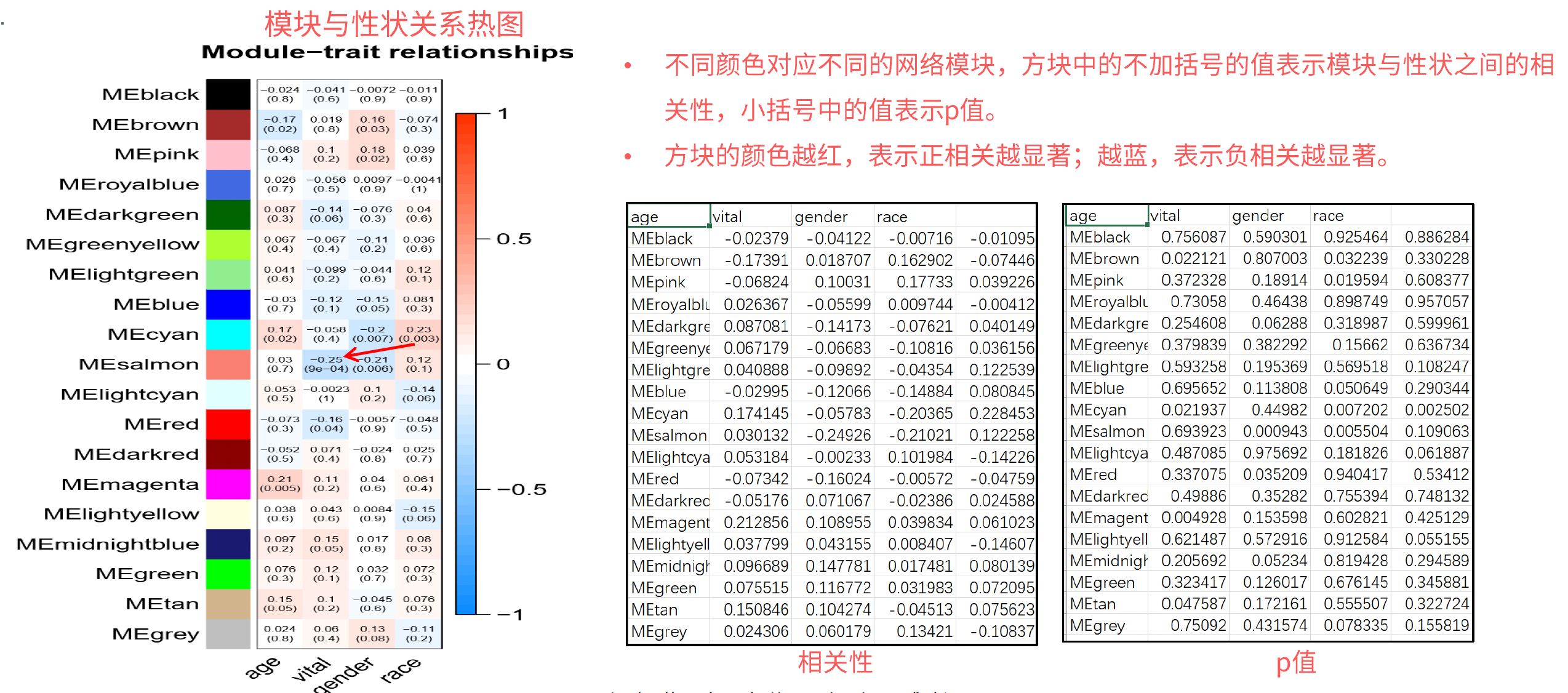
- GS: (Gene Significance) ,表示基因显著性,定义单个基因与外部信息的关联性,即基因与某个性状的相关性。
- MM: (Module Membership) ,用于描述模块的指标,是基于GS值计算出来。
- GS和MM高度相关, 与某个性状高度相关的基因所在的模块往往也与该性状相关。
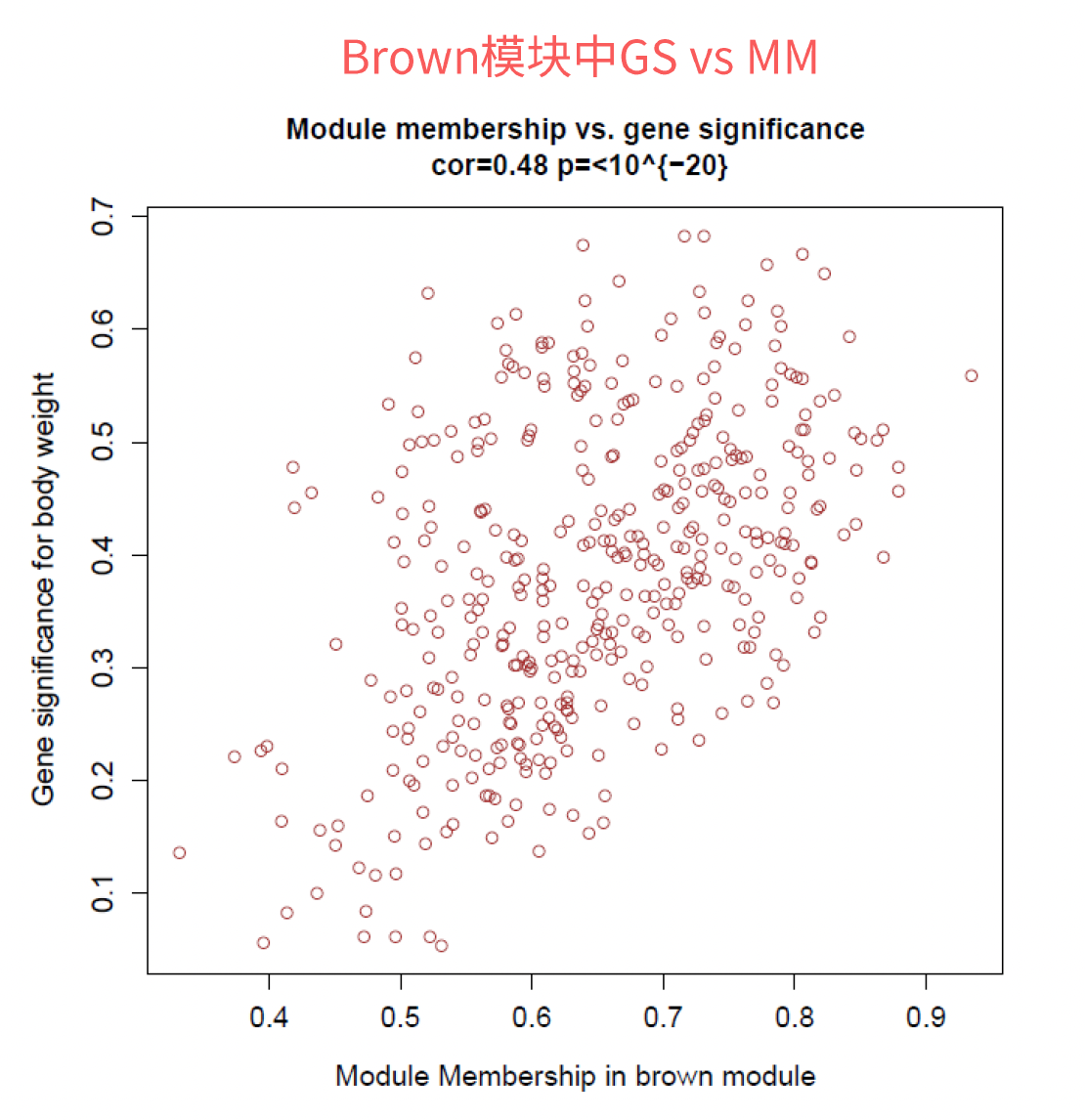
- 图中的横坐标表示MM值,纵坐标表示GS值,可以看出在brown模块中GS值和MM值高度关联,从p值看也是显著相关的。
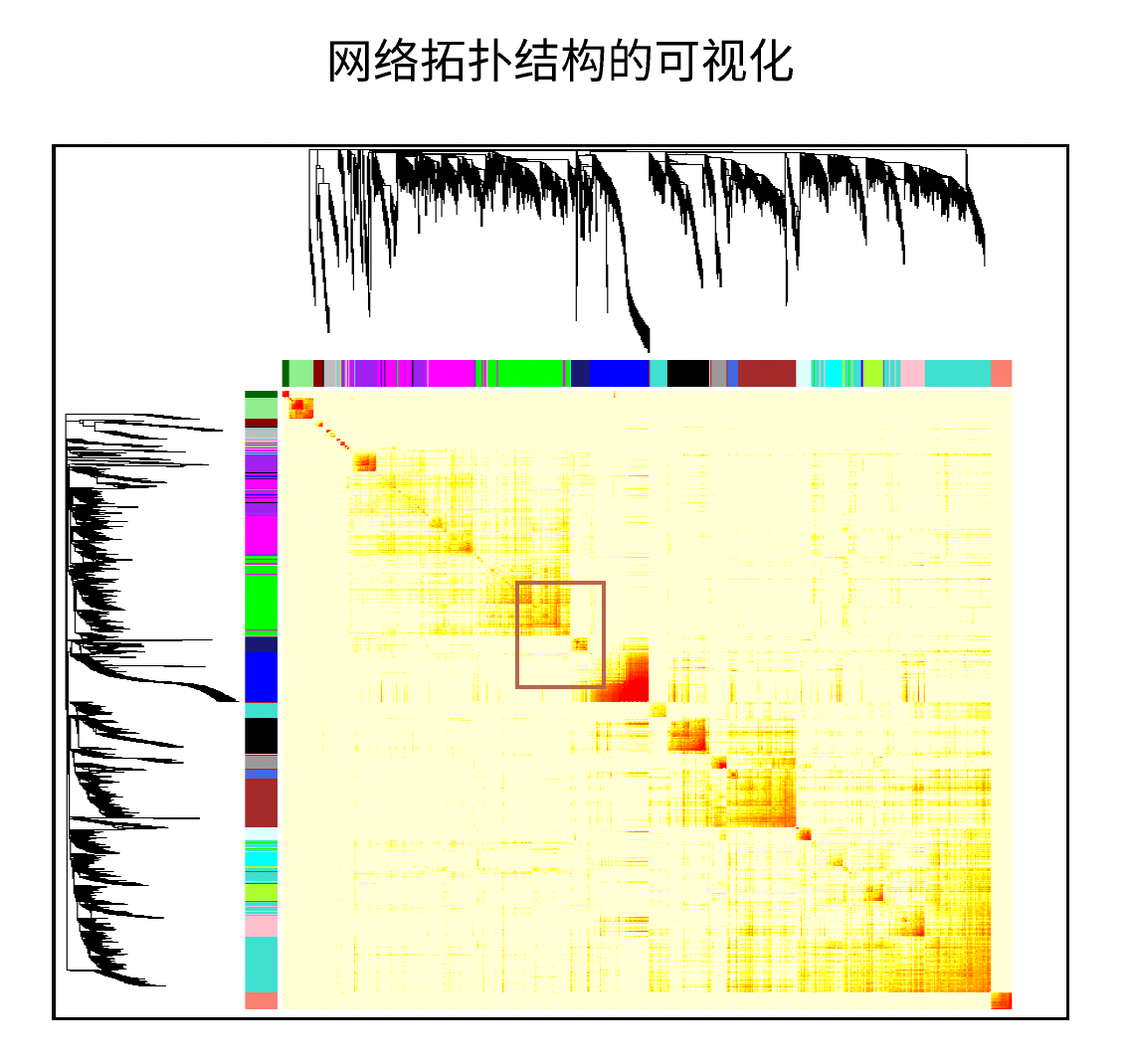
图中是对网络模块拓扑结构的可视化,使用的数据是网络拓扑(TOM)矩阵,上侧和左侧表示层次聚类结果。从热图可以看出,热图的结果与聚类的树枝一一对应。
评论
匿名评论隐私政策
✅ 你无需删除空行,直接评论以获取最佳展示效果











